Bessere Indikatoren für die Tumordiagnose
Biomarker sind für treffsichere Diagnosen oft unverzichtbar. Das Fraunhofer-Institut für Digitale Medizin MEVIS arbeitet an lernfähigen Algorithmen, die die Suche nach neuen Biomarkern deutlich erleichtern. Mediziner können dadurch wertvolle Unterstützung bei der Wahl der bestmöglichen Therapie erhalten.
Biomarker sind ein wichtiger Baustein der Diagnostik. Ein gängiges Beispiel ist der Cholesterinspiegel im Blut. Er kann auf ein erhöhtes Risiko für Herz-Kreislauf-Erkrankungen hindeuten. Auch in der Pathologie – der mikroskopischen Untersuchung etwa von tumorverdächtigen Gewebeproben – spielen Biomarker eine maßgebliche Rolle: Liegen bestimmte Zelltypen mit gewissen Eigenschaftskombinationen vor, lässt sich das als aussagekräftiger Indikator auffassen, der im Idealfall verrät, welcher Untertyp eines Tumors vorliegt. Dadurch können die Mediziner dann eine zielgerichtete, patientenindividuell wirksame Therapie wählen.
Nur: In der klinischen Praxis funktioniert dieses präzise Vorgehen längst nicht immer. So ergab eine Studie aus dem Jahr 2018, dass 44 Prozent der Krebspatienten in den USA für eine spezielle Art der Tumorbehandlung in Frage kommen sollten, die Immuntherapie. In der Realität schlug diese Behandlung dann aber nur bei 12 Prozent der Patienten an – viele wurden also vergebens behandelt. „Um künftig zielgerichteter therapieren zu können, müssten die Tumorarten noch viel genauer untergliedert werden können als bislang“, sagt MEVIS-Forscher Johannes Lotz. „Dafür müsste man jene Biomarker finden, die diese Unterscheidung erlauben.“
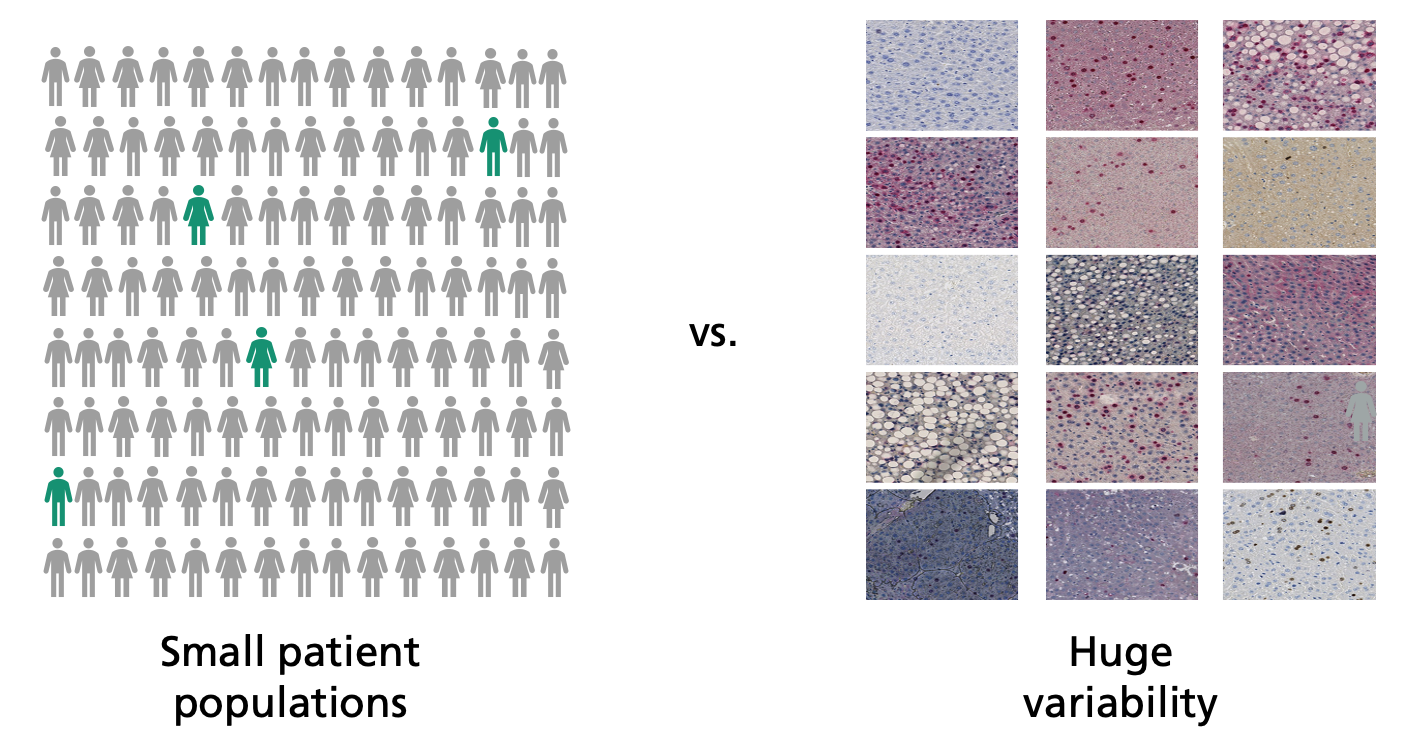
Um solche Biomarker aufzuspüren, braucht es aufwändige klinische Studien. In Zukunft sollen lernfähige KI-Systeme diese Suche unterstützen. „Der Computer analysiert digitalisierte Gewebeschnitte und fahndet darin nach Mustern“, beschreibt Lotz‘ Kollege Henning Höfener die Strategie. „Dadurch kann er neue Biomarker finden.“ Dafür muss die Software mit möglichst vielen hochwertigen Datensätzen trainiert werden, sonst kann die Mustersuche nicht gelingen. Hier taucht ein Problem auf: Je genauer man zwischen verschiedenen Untertypen eines Tumors unterscheiden möchte, umso weniger Patienten zeigen einen bestimmten Untertyp, und umso weniger Datensätze stehen für Training und Analyse zu Verfügung.
Das Beste aus zwei Welten
Eine weitere Schwierigkeit: „Die Arten der digitalen Gewebeschnitte können sich von Labor zu Labor deutlich unterscheiden“, erläutert Lotz. „Das erschwert es dem Computer, vorhandene Muster in den Bildern verlässlich aufzuspüren.“ Mit den bisherigen KI-Methoden, bei denen die Algorithmen Unmengen von Bildpunkten durchforsten, ist diesen Problemen kaum beizukommen. Deshalb versucht sich das MEVIS-Team an einer neuen Strategie und schaut sich Einiges von der bewährten Arbeitsweise des Menschen ab: „Erfahrene Pathologen haben Abertausende von Gewebebildern gesehen und daraus die wesentlichen Gesetzmäßigkeiten abgeleitet“, erklärt Höfener. „Anders als der KI genügt ihnen für eine treffsichere Diagnose dann meist eine Handvoll von Bildern.“
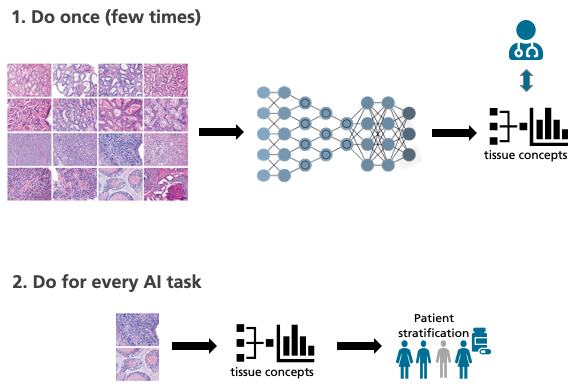
Das MEVIS-Team will das Beste aus beiden Welten miteinander verknüpfen. Der Plan: Die künstliche Intelligenz wird zunächst ähnlich trainiert wie ein Pathologe, und zwar mit vielen Gewebeschnitt-Bildern, die nichts mit einer speziellen Fragestellung zu tun haben. Durch diese „Grundausbildung“ eignet sich die KI allgemeine Merkmale und Gesetzmäßigkeiten an, sogenannte Gewebedeskriptoren. Mit deren Hilfe kann die Maschine die Bilder beschreiben und klassifizieren. „Setzt man dem Algorithmus dann eine konkrete Fragestellung vor, kann er anhand dieser Gewebedeskriptoren auch mit relativ wenigen Daten Korrelationen aufspüren, die zum Beispiel den Erfolg einer bestimmten Therapie vorhersagen“, sagt Höfener.
Noch steht das Projekt am Anfang. Doch Fraunhofer MEVIS besitzt gute Voraussetzungen, es erfolgreich zu meistern. „Wir haben große Erfahrung beim Trainieren lernfähiger Algorithmen und beim Programmieren von computergestützten Diagnosewerkzeugen“, betont Henning Höfener. „In Zusammenarbeit mit Projektpartnern aus der Klinik können wir die Daten auswählen, die wir für das Ersttraining unserer Algorithmen benötigen.“
KI beschleunigt automatische Erkennung
Von diesen Biomarker-Algorithmen sollen vor allem die Forschungsabteilungen von Pharmaunternehmen profitieren, aber auch Arbeitsgruppen an Universitäten. Doch der Ansatz verspricht weitere Anwendungen: So könnten sich die Gewebedeskriptoren für die Segmentierung nutzen lassen, also die automatische Erkennung und Vermessung von Gewebestrukturen in einem Bild. „Mit den Deskriptoren würden wir hier ebenfalls weniger Trainingsdaten brauchen“, erklärt Höfener. „Das könnte die Anpassung des Segmentierungsalgorithmus deutlich beschleunigen.“
Auch für die inhaltsbasierte Bildersuche (Content Based Image Retrieval) könnte die Methode hilfreich sein. „Das kennt man vielleicht von Google – man wirft ein Bild rein und bekommt dann ähnliche angezeigt“, erklärt Johannes Lotz. „Doch für medizinische Bilder funktioniert das noch nicht so gut.“ Das Deskriptoren-Konzept könnte deutliche Fortschritte bringen, hoffen die MEVIS-Fachleute. Die Vision: Bei einem ungewöhnlichen Befund könnten Mediziner in Datenbanken nach ähnlichen Bildern suchen, um ihre Diagnosen zu überprüfen und herauszufinden, welche Therapien in der Vergangenheit angeschlagen haben und welche nicht.
 Fraunhofer-Institut für Digitale Medizin MEVIS
Fraunhofer-Institut für Digitale Medizin MEVIS